(3)(4)(5)(6)(7)(8).jpg)
مراحل تهیه استرپتوکیناز :
1 – ژن تولید کننده این آنزیم در سویه هایی از استرپتوکوکها وجود دارد .
برای افزایش بازده تولید محصول سویه ای را انتخاب می کنیم که بیشترین مقدار استرپتوکیناز را تولید می کند . به این عمل micro organism screening(غربالگری ) گویند بدین منظور سویه های مختلف استرپتوکیناز را رشد می دهیم و سویه ای را انتخاب می کنیم که بیشترین و پر قدرت ترین استرپتوکیناز را تولید می کند .
2 – کشته سویه ی انتخابی استرپتوکوکها : چون نیاز به مقدار زیادی از DNA است لذا سویه ی انتخابی را در محیط کشت رشد می دهیم تا PLATE سلولی تشکیل شود. ( منظور مجموعه سلولی است )
2 نوع محیط کشت داریم :
A : Defind medium مثل M9
B : Complex or undifind medium مثل LB (luria-bertani)
LB هم 2 نوع است:
1 – Brows LB : به فرم مایع است
2 – LB Agar : به فرم جامد است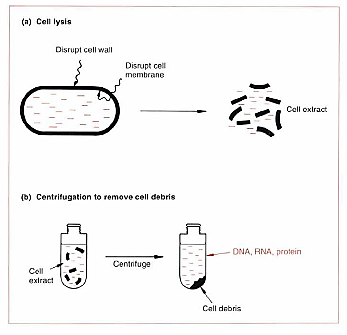
مقایسهA وB:
A : محیط defind :
I - نوع و مقدار هر جزء در محیط مشخص است ( مثلاً مقدار و نوع aa یا نوع منبع کربن و انرژی مشخص است )
II - همیشه این محیط کشت استفاده نمی شود چون گران است
III - اگر بخواهیم M.O[5] در شرایط کنترل شده و خاص کشت شود از این محیط استفاده می شود .
B : محیط undifind :
I -نوع و مقدار هر جزء مشخص نیست
II - ا ساس آن عصاره مخمر ( برای مواد غذایی Nutriants و یونهای Na+ ، k+ ، Mg2+ ) و تریپون ( منبع تولید آمینه اسیدهای مختلف ) است .
برای کشت و تکثیر سویه انتخابی استرپتوکوک از LB brows استفاده میشود . پس از کشت harvesting می کنیم یعنی با سانترفیوژ سلولها را از بقیه اجزاء محیط کشت جدا می کنیم
3 – جدا کردن DNA از کل سلول ( Cell extract) :
باید سلول را بشکنیم. به 2 طریق امکان پذیر است:
A – فیزیکی و B – شیمیایی
AM – فیزیکی : از نیروهای فیزیکی استفاده می شود مثلاً Sonication به کمک sonicator
BM – شیمیایی :
1 - اغلب در آزمایشگاهها از این روش استفاده می شود
II -مثلاً برای استریتوکوک از EDTA ؛ SDS لیزوزیم استفاده می شود بدین طریق ساختار پلی مری پیتدوگلیگان دیواره سلولی باکتری شکسته می شود.
EDTA G[6] :
یک Shelating agent است یونهای دو ظرفیتی (خصوصاً +2Mg ) را به خوبی جذب می کند سبب تغییر در integrity و استحکام cell wall می شود و با جذب
Mg 2+ آنزیمهای DNAase و RNAase که پس از شکسته شدن MO آزاد شده اند را
غیر فعال کند, چون اسید و آنزیم برای فعالیت، Mg2+ لازم دارند
Aلیزوزوم :
سبب تخریب Cell well می شود .
SDS[7] B :
بر خلاف EDTA و لیزووزرم Call wall را تخریب نمی کند بلکه روی غشای سلولی اثر دارد با نفوذ در بین ساختار فسفولیپیدی غشاء با خاصیت دترژانتی خود سبب به هم ریختگی کامل ساختار غشاء می شود .
Note ( : کلاً با EDTA و لیزوزوم به دلیل فروپاشی Cell wall و به دلیل اسمولاریته موجود در محیط خود بخود سلول می ترکد و نقش SDS به عنوان کمک کننده است . حال در محیط Cell debris ( باقی مانده سلولی ) RNA ، DNA و پروتئین داریم .
4- جدا کردن DNA از [8]Contaminants ( Purification : خالص سازی ) :
به 2 روش انجام می شود:
A : دگرده کردن Contaminants
B : Ion exchange chromatography
5note : قبل از شروع Purification به کمک سانترفیوژ ساده مخلوط پروتئین ، DNA ، RNA را از Cell debrize (چون دانسیته بالایی دارند ته نشین می شوند ) جدا می کنیم
.
: دگرده کردن A : Contminants
از سیستم فنول کلروفرم استفاده می کنیم پروتئینها به راحتی در فنول حل می شوند وقتی محیط با نسبت 1 به 1 فنول کلرو فرم استفاده کنیم, شاهد 2 فازه شدن Cell extrant در دو فاز آبی و آلی فنول کلروفرم خواهیم بود بدین صورت که پروتئین هایی که در
اثر حل شدن دناتوره شده اند بین دو فاز مایی و آلی قرار می گیرند (درمرز) و محیط بالای مرز که فاز مایی است متشکل از DNA , RNA استïپروتئینها جدا شدند
مرحله بعدی جدا کردن RNA و DNA است ï از ریبونوکئاز ( RNAase ) استفاده می کنیم
مزیت : خیلی به صورت روتین این سیستم در آزمایشگاهها به کار می رود چون کم هزینه است
عیب : قدرت خالص سازی پایینی دارد .
B : ION EXCHANGE CHROMATOGRPHY:
-1 ستون که حاوی بستر رزین با بار + است
2 - RNA و DNA زنجیره پلی نوکلئوتیدی منفی هستند ولی DNA ازRNA منفی تر است چون قنددئوکسی ریبوز دارد .
3 - بار پروتئینها فقط در PH (ایزواکتریک ) صفر است
با ریختن مخلوط حاوی DNA و RNA ،PRO ,آنهائی که بار منفی دارند به ستون می چسبند .برای جدا کردنشان، از محلولهای نمکی استفاده می کنیم. هر چقدر غلظت یونهای نمکی بالاتر باشد قدرت بیشتری را برای شکستن پیوند بین مولکول متصل به رزین را دارند و خود در رقابت جایگزین مولکول می شوند .
مرحله اول : که غلظت محلول نمکی کم است پرتئینها جدا می شوند
مرحله دوم : جدا شدن RNA
مرحله سوم : غلظت محلول نمکی بالاست لذا DNA جدا می شود
استخراج پلاسمید :
DNA کرموزومی را برای استخراج ژن لازم داشتیم و پلاسمید را برای ساخت وکتور لازم داریم.
وقتی کروموزوم سلول را جدا می کنیم یعنی غلظتی از DNA و پلاسمید را داریم که برای جدا کردن ایندو باید ببینیم چه تفاوتهایی با هم دارند :
1 – سایز : DNA کروموزومی خیلی بزرگتر است
2 – Conformation
#جداسازی DNA از پلاسمید بر اساس اختلاف در سایز:
1 - وقتی به محیط EDTA و SDS و لیزوزوم اضافه می کردیم سلول میترکید وDNA متصل به غشاء تکه تکه میشد. با اضافه کردن سوکروز به محیط ،محیط هیپرتونیک می شود (افزایش غلظت ) و دیگر سلول نمی ترکد غشاء بازشده در پی جداشدن cell debrise، عده زیادی از DNA کروموزومی هم با آنها خارج می شود .
2- استفاده از 100 X trition به جای SDS : تریتون 100 X یک دترژانت است عامل متلاشی کردن غشاء است اما به خاطر غیریونی بودنش DNA را نمی شکند.
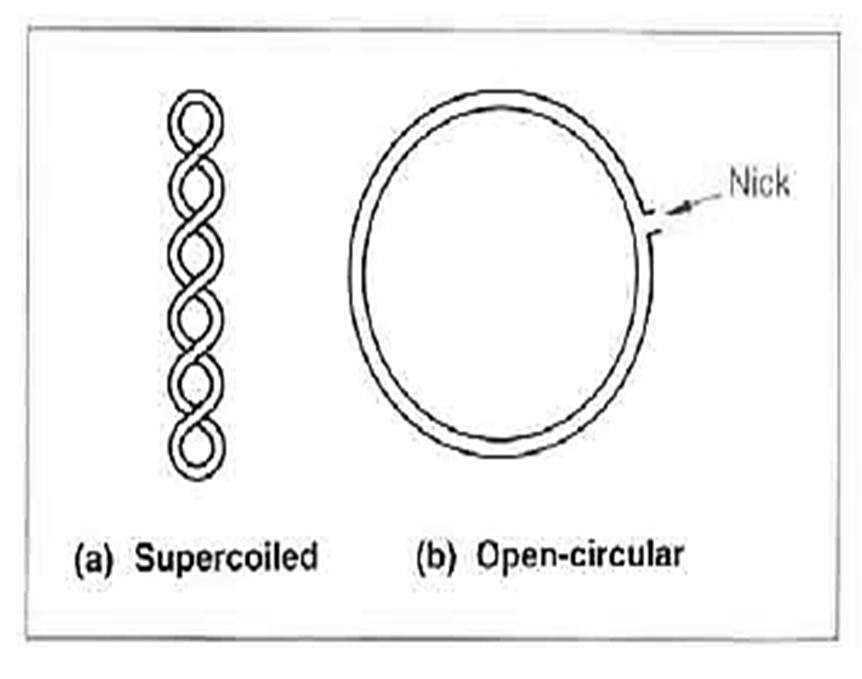
# 2 – جداسازی براساس Conformation :
شباهت DNA و پلاسمید : هر دو حلقوی ، بسته و Supercoil ( به دور خود بپیچد ) هستند.
در پلاسمید اگر بین 2 نوکئوتید آن شکافی ایجاد شود از حالت SUPERCOIL خارج شده و به فرم Relax تبدیل می شود .
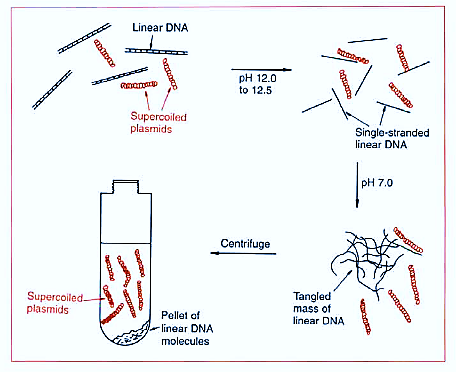
برای کار بیوتک نیاز به پلاسمیدی است که Supercoil باشد بعد از گذشت مدت زمانی از کشت ,MO پلاسمید به این فرم است این زمان در انتهای فاز لگاریتمی است در این حالت میتوان پلاسمید را از DNA جدا کرد چون DNA به صورت خطی و خرد شده در آمده است. لذا PH را به 5/12 – 12 می رسانیم این PH سبب جدا شدن 2 زنجیره DNA خطی می شود اما پلاسمیدی که سوپر کویل است چون خیلی 2 زنجیره به هم نزدیک هستند لذا پیوند هیدروژنی بینشان شکسته نمی شود .
بعد محیط را اسیدی می کنیم سبب Renacharing و تشکیل پیوند هیدروژنی بین قطعات DNA می شود اما به صورت اگریگه اتفاق می افتد چون بدون قانون است و سوپرکویل در محیط اسیدی تغییر نمیکند لذا DNA اگریگه شده به علت دانسیته بالای خود از پلاسمیدهای سوپر کویلی به کمک سانترفیوژ قابل افتراق هستند
دفتر مرکزی
کرج - بعد از پل آزادگان - ابتدای خیابان مطهری- نبش کوچه ساوجی - ساختمان کوروش پلاک 901 - طبقه اول واحد 1- کد پستی: 3149649837
تلفن های تماس
026-32555937 - 026-32555938
09363928058 - 09123050747
09120796671 - 09380626117
asadimohammadreza@yahoo.com
kalantari.ali67@gmail.com
.png)